

La Génétique
Le Génome
Un point d’Histoire :
Les premiers outils permettant d’analyser la molécule d’ADN, donc les gènes, sont apparus à partir de 1975. En 1985, l’idée d’un programme spécifique au séquençage du génome humain « le projet génome humain » est lancé. Il fut entrepris dès 1989 sous la direction du « Human Genome Organisation » (HUGO). C'est ainsi qu'en l'an 2000, Bill Clinton annonce que le séquençage brut du génome humain est achevé. On pensait alors que le séquençage de la totalité du génome aurait permis de comprendre la totalité de la nature humaine, ce qui évidemment, est faux. Les gênes expliquent en effet une grande partie des individus, mais ne font pas tout (environnement et épigénétique).
En 2007, de nombreuses recherches ont porté sur des études comparatives du génome entier : les chercheurs ont comparé les ADN de milliers d'individus malades et sains afin d'identifier les variantes génétiques légères associés à un risque plus élevé d'avoir la maladie étudiée. Ainsi, on ne connaît la signification que de 3% du génome, de surprise en surprise, les généticiens se rendent compte que l'ADN est d'une complexité étonnante, exemple récent avec la preuve de l'existence in vivo des quadruplex, structure de l'ADN nouvelle pour les généticien. Seulement 25000 gênes codent une protéine, alors qu'on s'attendait à plus.
Ces dernières années, avec la découverte progressive des gènes responsables des maladies héréditaires, des possibilités de diagnostiques ont été offertes pour un nombre croissant de maladies. Il reste encore beaucoup de travail puisque, actuellement, cette approche ne concerne qu’un peu plus d’une centaine de maladies alors qu’il en existe plusieurs milliers. Pour ce champ de la génétique, imaginer ce que sera le futur dans les vingt ans à venir est simple. Maintenant que la séquence du génome humain est connue, les gènes responsables des maladies héréditaires vont être progressivement découverts. Après chaque découverte, il sera possible de proposer une analyse du gène chez chaque malade concerné. Ainsi il pourrait être possible à l'avenir de connaître l'exacte expression de chacun des gênes, et toutes leur allèles, ainsi les diagnostiquer.
Le Génome, qu'est-ce que c'est ?
Le génome est souvent comparé à une encyclopédie dont les différents volumes
seraient les chromosomes. Les gènes seraient les phrases contenues dans ces
volumes et ces phrases seraient écrites dans un langage génétique représenté
par quatre bases (adénine, guanine, cytosine et thymine) abrégées en AGCT.
La molécule d'ADN est une macromolécule organisée en double hélice,
avec une complémentarité des nucléotides A, T et C, G ;
pour adénine, thymine, guanine, et cytosine.
Le génome est l'ensemble du matériel génétique d'un individu ou d'une espèce,
codé dans son ADN. Il contient en particulier toutes les séquences codantes
(transcrites en ARN messagers, et traduites en protéines) et non-codantes
(non transcrites, ou transcrites en ARN, mais non traduites).
Chez l'humain (organisme eucaryote), le génome nucléaire est réparti sur
46 chromosomes, soit 22 paires d'autosomes (semblables) et deux gonosomes
(chromosomes sexuels) (XX chez la femme, XY chez l'homme).
Le nombre des gènes dans le génome des organismes vivants varie beaucoup moins que la taille du génome. Chez la plupart des organismes vivants il est compris entre 1 000 et 40 000. les génomes humains sont identiques à 99.9 %, avec seulement 100 000 nucléotides qui diffèrent les uns des autres.
Chez l’homme le nombre de gènes tournent autour de 30 000 gènes et son génome est de la taille de 3400,0 Mpb soit 3.400.000.000 paires de bases. 200 de ces gènes sont influençables par le mode de vie.
« Gattaca » est une fiction qui développe les résultats d'une société qui pratiquent une forme d'eugénisme. La technologie y est plus avancée qu'aujourd'hui, et des résultats tels que ceux-ci font rêver nombres de généticiens :
Le génome est donc un inventaire très complexe et bien rempli des gênes à l’origine des caractères humains. Son étude permettra de diagnostiquer des maladies sur des embryons, au cours de diagnostique prénatals. Mais, seulement ?
Si les connaissances en termes de génome permettent de comprendre la signification de certains des gênes, et dans l'absolu de l'ensemble du patrimoine génétique, le séquençage quant à lui permet de connaître, pour chaque individu, les séquences de nucléotides complètes.
Le Séquençage
Un point d'Histoire :
Les fondements du séquençage ADN sont relativement récents au regard de l’histoire de l’humanité. En voici les principales étapes :
Dans la seconde moitié du 19ème siècle, le biochimiste suisse Friedrich Miescher isole une substance inconnue jusqu’alors. Il démontra qu’elle contenait du phosphore, élément nouveau pour les chercheurs, et qu’elle était acide. Elle fut nommée acide désoxyribonucléique (ADN).
Le britannique William Astbury étudie l’ADN dans les années 1930. Il démontre sa structure : long filament, et sa composition : succession de bases empilées selon un espace régulier de 0.34 nm. Quatorze ans plus tard, Oswald Avery démontre que l’ADN se transmet d’un individu à l’autre.
Dans les années 1960 le fonctionnement interne global de l’ADN était connu. Les chercheurs estimèrent le remplacement des gènes défectueux théoriquement possible. Une technique capable d’intervenir sur les molécules fut mise au point.
Ce fut la découverte des enzymes dites endonucléases de restriction en 1965 par Werner Arber, Daniel Nathans et Hamilton Smith qui rendit opératoire la biologie moléculaire. Ainsi le séquençage des gênes devenait réalisable. En 1971, la première séquence comportant un gène étranger est créée par Paul Berg. Dans les années 1970, la législation évolue vers un contrôle plus strict, les autorités se rendant compte des risques que comportait lé génétique.
En 1983, Kary Mullis invente une méthode, la PCR (polymérase chain reaction). En la combinant avec la Taq polymérase (ADN polymérase stable à haute température), on fut en mesure d’identifier une personne à partir de ses empreintes génétiques. Cette méthode fut introduite en médecine légale à partir de 1987. Cette technique amplifie les fragments d'ADN pour ensuite les étudier... en effet le matériel génétique doit être assez conséquent pour ce faire, mais les progrès limitent cette nécessité.
Dans cet extrait du film « Bienvenue à Gattaca », un séquençage est pratiqué très rapidement sans passer par une amplification apparente :
Dans cet extrait de « Gattaca », il semble qu'un seul cheveu suffira à l'avenir pour séquencer un génome... Sceptique ? Rendez-vous à la fin de cette partie...
En 1980, Olson et David Burke clonèrent une grande quantité d’ADN (250kb) et construisirent des chromosomes artificiels de levure.
A partir de 1990, le séquençage complet du génome de plusieurs espèces fut entrepris :
• Du virus
• De bactéries pathogènes ou non
• Du Nématode Coenorhabditis elegans (sorte de vers)
• De la drosophile (une mouche)
• De la plante Arabidopsis thaliana
En 1985, l’idée d’un programme spécifique au séquençage du génome humain est lancée. Il fut entrepris dès 1989 sous la direction du Human Genome Organisation (HUGO). Treize ans plus tard, moyennant 3 milliards de dollars, alors que l'on croyait tout expliquer par le patrimoine génétique, Bill Clinton annonce que le séquençage brut du génome humain est achevé.
Le séquençage, Qu'est-ce que c'est ?
Le séquençage de l'ADN consiste à déterminer l'ordre des
chaînes de nucléotides pour un fragment d’ADN donné.
C’est une technique souvent utilisée dans les laboratoires
de biologie. Cette technique utilise les connaissances qui
ont été acquises sur les mécanismes de la réplication
de l’ADN depuis une trentaine d’années.
L’ADN est une longue molécule qui contient l’information
génétique d’un individu. Il est situé dans le noyau des
cellules et son appellation ADN signifie
"acide désoxyribonucléique". Il a la forme d’une
double hélice et forme de longues chaînes de
matériel génétique organisé en chromosomes.
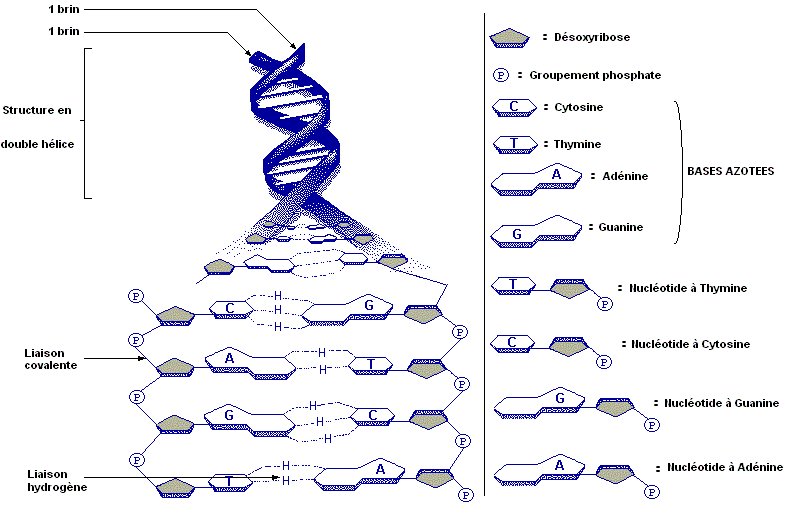
Un brin d’ADN est composé d’une chaîne de
molécules appelées nucléotides. Un nucléotide,
c’est le groupement d’un sucre désoxyribose,
d’un groupement phosphate et d’une base azotées.
Il y a 4 bases azotées :
A (adénine), T (thymine), C (cytosine), G (guanine).




Un caryotype complet
Organisation en double hélice
et complémentarité des bases
azotées de la molécule d'ADN
Transcription et traduction au sein de la cellule
Localisation de la molécule d'ADN dans les chromosomes

Séquenceur "Ion proton"
Aujourd'hui le séquençage a franchi un nouvel horizon, celui de l’accessibilité maximale. En effet, depuis peu, des séquenceurs de la taille d'une imprimante laser, du prix d’une voiture sont vendus sur les marchés, et peuvent être achetés par des particuliers, ou de petits laboratoires privés.
Exemple très concret avec le « Ion proton » de l'entreprise Life Technologies.
Le prix par génome séquencé est de seulement 1000$. Pour ce prix,
l'analyse du génome est aussi offerte, au regard des limites qu'imposent
les connaissances limitées certes, mais des caractères ou des prédispositions
sont ainsi évaluées. Ainsi la revue « Geeko » affirme :
« Il faut reconnaître que de nombreux petits laboratoires,
spécialistes et même amateurs seront tentés de mettre la main dessus.»
Et,
« Ce séquenceur ADN ouvre la recherche à tout un chacun. »
Il serait alors possible de séquencer un génotype entier, puis l'analyser, c'est à dire comprendre quel gêne est lié à quel caractère (ex: couleur des yeux, forme du visage, probabilité de devenir diabétique et tant d’autres..) pour l'individu en question. En terme de technologie toujours, des clefs USB contenant l'ensemble des informations génomique ont vues le jour, chez Knome (The Human Genome Interpretation Company). D'autre part, le séquençage personnalisé se développe fortement surtout aux États-Unis, ou de nouvelles firmes se développent, ainsi "Illumina", "23 ans me", "deCODEme", ou "Navigenics" proposent à des prix attractifs des séquençage et leur interprétations. (liens des sites dans l'annexe)
Toujours aussi sceptique ?
À l'avenir, on pourrait imaginer de tels résultats, avec un effort combiné d'un séquençage ultra performant, et d'une connaissance du génome parfait : (Extrait du film de fiction « Gattaca », alors vrai ou fausse fiction ? Nous allons essayer de décrypter les mécanismes scientifiques et génétiques d'aujourd'hui).
Un séquençage et une analyse complète d'un génome de 3milliards d'éléments en 4 secondes, au service du statut de « Valid »
Qu'est-ce que ce genre de nouvelles technologies représente concrètement ? Liée à la connaissance du génome, que pourrait-on imaginer faire avec l’avancée de ce domaine ?
Le DPI va de pair avec la connaissance du génome. En effet on l'utilise lorsque l'on veut repérer des caractères chez l'embryon, il faut ainsi procéder à un diagnostique.
...


Le Diagnostic Préimplantatoire
Le DPI, Qu'est-ce que c'est ?
Le diagnostic préimplantatoire consiste à rechercher une maladie génétique grave et incurable, qui peut être transmise par les parents sur des embryons conçus in vitro c’est-à-dire « dans une éprouvette » à l’aide des spermatozoïdes du père et des ovules de la mère. Cet examen est pratiqué à titre exceptionnel. Il vise à replacer dans l’utérus de la mère, un embryon ne portant plus la maladie. Plus largement, il vise à repérer des caractères sur les embryons et à les selectionner pour obtenir celui qui correspond le mieux aux attentes. Après trois jours de développement, on brise la paroi à l'aide d'un laser, puis par aspiration, on prélève une ou deux cellules de l'embryon qui en possède huit. Par suit, on procède à une PCR qui répliquera les brins d'ADN un grand nombre de fois, ou à un FISH qui localise les séquences « anormales » des gênes. Aujourd'hui les séquençages plus performants donnent de nouveaux espoirs en terme d'efficacité et de possibilités.
Un point d'Histoire :
En 1987, le DPI apparaît dans la presse. Trois ans plus tard, le premier DPI est réalisé en Angleterre avec la détermination du sexe de l’embryon pour une maladie présente uniquement chez un garçon qui naîtra deux ans après, en 1992. En France, il attendre 1994 pour la légalisation, et 2001 pour la naissance du premier garçon, Valentin à l’hôpital Antoine Bèclére.
Aujourd'hui les DPI et les FIV (fécondations in Vitro) sont de plus en plus courantes, certains couples demandent aux médecins capables de les réaliser des enfants aux capacités améliorées... à l'image de « The Fertility Institue en Californie (développée en partie 2).
Ainsi le diagnostic préimplantatoire examine et repère les caractères sur un embryon, pour favoriser son apparition, ou pour tenter de l'éviter. À son service, le séquençage, lié à la connaissance du génome, qui permettent la mise en évidence d'une palette toujours plus grande de caractères. Les enzymes de restriction quant à elles, pourraient agir sur son information génétique... Quels espoirs, quelles craintes est-il légitime d'avoir au regard des applications de ces techniques ?
Ici, dès la naissance de l'enfant, on connaît déjà tout de lui...
Un enzyme restrictive à la différence d'une séquence est un complexe de protéine qui permet de couper la molécule d'ADN à différents endroits, ainsi on peut procéder à des mutations artificiels.
Les Enzymes Restrictives
Une enzyme de restriction, Qu'est-ce que c'est ?
Les enzymes de restriction sont une sorte de ciseaux
moléculaires, c’est une protéine pouvant cliver (fendre) un
fragment d’ADN au niveau d’une séquence de nucléotides
bien précise appelée "site de restriction". Chaque enzyme a
un site de restriction prédéfini. Elles sont devenues des outils
important dans la génétique.
- Différents types d'enzymes :
Enzymes de type I : Ces enzymes reconnaissent un site particulier qui n’a aucune symétrie, qui ne coupe pas l’ADN à ce niveau mais à environ 1000 jusqu’à 5000 paires de bases plus loin et libère plusieurs dizaines de nucléotides.
Enzymes de type II : Ces enzymes sont les plus nombreuses et les plus utilisées aux laboratoires de génie génétique. Leurs sites de restrictions de 4 à 10 paires de bases sont des séquences palindromiques (se lisent de droite à gauche et inversement, comme le mot radar et la phrase "esope reste ici et se repose"). Elles coupent au niveau de leurs sites de restriction.
Enzymes de type III : Ces enzymes reconnaissent une séquence de nucléotides mais coupe à une vingtaine de paires de bases plus loin.
Il y a un nombre indéfini d’enzymes de restriction, on en connaît aujourd’hui plusieurs centaines.
- Différentes coupures :
Lorsqu’une enzyme de restriction clive un ADN, il y a deux possibilités :
- Soit une coupure franche c'est-à-dire une coupure verticale au même niveau sur les deux brins. Ces coupures sont appelées les bords ou les bouts francs : la coupure a lieu au milieu du site de restriction. Il ne peut pas y avoir de liaison spontanée entre les fragments issu de cette coupure. Seule une T4 ligase peut coller deux brins d’ADN issue d’une coupure franche.
- Soit une coupure cohésive, c’est-à-dire que les séquences nucléotidiques sont identiques sur les deux brins mais en orientations antiparallèles. sur les deux brins. Les bouts des deux ADN obtenus ne sont plus francs ; ce sont des bouts collants ou bouts cohésifs. Ces enzymes sont palindromiques. Les parties simples de ces brins peuvent s’apparier, d’où la possibilité de lier des fragments d’ADN d’origine différentes : c’est le phénomène de la recombinaison génétique.
Dans cette petite animation est représentée l'action de l'enzyme Ecor1 sur la molécule d'ADN...
Nous avons aussi procédé à un TP sur le logiciel "Anagène" pour comprendre le fonctionnement des enzymes de restrictions, nous avons chercher les enzymes appropriées pour trouver à quel allèle, l'allèle à tester s'identifiait :
- Histoire des enzymes :
La découverte des enzymes dites endunocléases de restriction en 1965 par Werner Arber, Daniel Nathans et Hamilton Smith rendit opératoire la biologie moléculaire.
Cette découverte des enzymes de restriction découle essentiellement aussi d’observations faites sur le développement de bactériophages (virus affectant que les bactéries) dans les années 1960.
- Nomenclature des enzymes de restriction :
En 1973, Smith et Nathan ont proposé une nomenclature qui est définitivement adoptée. Chaque endonucléase a un nom de code déterminé selon les principes suivants :
- La première lettre en majuscule est l'initiale du genre de la bactérie.
- Les deux lettres suivantes, en minuscule désignent l'espèce de la bactérie.
- La quatrème lettre en majuscule, (pas toujours présente) désigne la souche bactérienne.
- Un chiffre romain distingue les enzymes d'une même souche dans l'ordre de leur découverte.
Exemple : EcoR 1:
E = Escherichia
Co = espèce Coli
R = Souche RY13
I = 1ère endonucléase isolée
- Isoschizomères et enzymes compatibles :
On appelle isoschizomères 2 enzymes différentes qui reconnaissent le même site de restriction.
Exemple :
Msp I : NNC/CGGNN et Hpa II : NNC/CGGNN
NNGGC/CNN NNGGC/CNN
Deux enzymes compatibles ont des sites de restriction différents mais donnent naissance aux mêmes extrémités cohésives.
C'est le cas de BamH I et Sau3AI qui donnent les mêmes extrémités cohésives car le site de reconnaissance de Sau3A1 est contenu dans celui de BamH 1.
Exemples :
BamH 1 : NNNG/GATCCNN et Sau 3A 1 : NNN/GATCNNN
NNNCCTAG/G NNNCTAG/NNN
Ainsi, BamH I donne les mêmes extrémités cohésives que Sau3A I avec qui elle est compatible.


Coupure franche
Coupure cohésive


Différentes enzymes de restrictions

- Schéma explicatif de l'ADN :
- Liaisons phosphodiester :
Dans les acides nucléiques, les différents nucléotides sont placés bout à bout et liés les uns aux autres par des liens 5’- 3’ (prononcé 5 prime – 3 prime) phosphodiester (PO4) : ces chiffres donnent le sens de la liaison :
5' - Nucléotide 1 - PO4 - Nucléotide 2 - PO4 - ... - 3'.
Le phosphate se lie au carbone 3 du sucre du premier nucléotide et au carbone 5 du sucre du nucléotide suivant ; tout ceci par l'intermédiaire de deux liaisons ester. Les liaisons phosphodiester sont des liens covalents. Le phosphate est donc le lien entre chaque sucre.
- Une liaison hydrogène est une liaison de faible énergie entre deux atomes attirés l’un vers l’autre pour des raisons électrostatiques, l’un étant riche en électrons donc nucléophile (« qui aime les charges positives ») et l’autre n’ayant que les protons de son noyau donc électrophile (« déficient en électrons»).
- Ainsi l’atome d’hydrogène, dont l’unique électron est par nécessité dans l’orbitale qui unit cet atome au reste de la molécule, ne peut qu’être électrophile et donc être attiré par les atomes ayant des doublets électroniques libres.
- Les atomes d’azote et surtout d’oxygène possèdent respectivement deux et quatre électrons de leur couche périphérique qui ne participent pas aux orbitales des liaisons covalentes de la molécule. Ceci leur confère un caractère nucléophile qui leur permet d’exercer une attraction sur les atomes électrophiles voisins, en particulier les atomes d’hydrogène : cette attraction constitue une liaison hydrogène.
Dans le cas de l’ADN, les deux brins sont disposés de telle sorte que toutes les bases azotées se retrouvent au centre de la structure. Cette structure appelée double hélice est maintenue par des liaisons hydrogène qui se forment entre les bases azotées complémentaires. L'adénine s’associant toujours avec la thymine (dans l'ADN) ou l’uracile (dans l'ARN) à l’aide de deux liens hydrogène et la guanine s’associant toujours avec la cytosine à l’aide de trois liens hydrogène. Les liaisons hydrogènes sont des liaisons faibles que la cellule peut aisément défaire.
On pourrait donc, grâce aux enzymes de restriction procéder à des mutations artificielles sur la molécule d'ADN opérées par l'homme, et grâce à l'avancée des connaissances d'une part sur le génome, d'autre part sur le séquençage, les enzymes de restriction prennent tout leur sens alors existe-t-il des contrôles ? Peut-on réellement agir sur la molécule d'ADN en toute liberté, qu'est-ce que cela induit en termes d'applications ?
...